Die Rechercheplattform PubPharm
Christina Draheim 1Kristof Keßler 1
Janus Wawrzinek 2
Stefan Wulle 1
1 Technische Universität Braunschweig, Universitätsbibliothek, Braunschweig, Deutschland
2 Technische Universität Braunschweig, Institut für Informationssysteme, Braunschweig, Deutschland
Zusammenfassung
Die Universitätsbibliothek Braunschweig betreut den DFG-geförderten Fachinformationsdienst (FID) Pharmazie in Kooperation mit dem Institut für Informationssysteme (IfIS) der Technischen Universität Braunschweig. Zentraler Dienst ist die pharmaziespezifische Rechercheplattform PubPharm (https://www.pubpharm.de/), ein auf der Open Source Software VuFind basierendes Discovery System. Aus der Zusammenarbeit mit dem IfIS resultieren innovative Suchfunktionen. PubPharm enthält auf die Bedarfe der universitären pharmazeutischen Wissenschaftler*innen zugeschnittene Inhalte und Funktionalitäten, die auch für andere Lebenswissenschaften interessant und nützlich sein können.
Schlüsselwörter
Pharmazie, Fachinformationsdienste, PubPharm
Der Fachinformationsdienst Pharmazie
Die pharmaziespezifische Rechercheplattform PubPharm (https://www.pubpharm.de/) ist der zentrale Service des Fachinformationsdienstes (FID) Pharmazie [1]. Dieses Kooperationsprojekt zwischen der Universitätsbibliothek Braunschweig und dem Institut für Informationssysteme (IfIS) der Technischen Universität Braunschweig wird seit 2015 von der Deutschen Forschungsgemeinschaft (DFG) im Förderprogramm „Fachinformationsdienste für die Wissenschaft“ unterstützt [2].
Ziel dieses Projektes ist es, die Literatur- und Informationsversorgung für die universitäre pharmazeutische Forschung signifikant zu verbessern, was die Weiterentwicklung der Informationsinfrastruktur, die Erforschung und Implementierung innovativer Recherchedienste und die Verbesserung des Volltextzugriffs umfasst [1].
Die Universitätsbibliothek Braunschweig betreute bis 2014 als Vorgängerprojekt das DFG-geförderte Sondersammelgebiet Pharmazie, mit dem die größte pharmazeutische Literatursammlung in Mitteleuropa aufgebaut wurde [3]. Erste Schritte zur digitalen Transformation erfolgten mit der Virtuellen Fachbibliothek Pharmazie ab dem Jahr 2000. PubPharm bietet in der Nachfolge einen Funktionsumfang auf einem weitaus höheren Niveau mit einer höheren Qualität der angebotenen Services. Der Mehrwert von PubPharm liegt inhaltlich in einem umfassenden Nachweis von zusätzlichen fachspezifischen Ressourcen, der über die im Fach etablierten Datenbanken hinausgeht.
Mit Unterstützung des Kompetenzzentrums für die Lizenzierung elektronischer Ressourcen (KfL) lizenziert der FID Pharmazie für die universitäre pharmazeutische Forschung spezialisierte Fachzeitschriften, die zuvor nicht flächendeckend an den 22 Pharmazie-Universitätsstandorten abonniert wurden und nicht zum Grundbedarf der universitären Literaturversorgung zählen [1]. Über diese sogenannten FID-Lizenzen erhalten die Wissenschaftler*innen unmittelbaren Volltextzugriff auf aktuell über 50 Zeitschriften von sechs Verlagen. Ergänzend dazu kann ein Zugriff auf die lizenzierten Zeitschriften an eine begrenzte Anzahl an Einzelnutzer*innen aus angrenzenden Fachbereichen vergeben werden. Die Zeitschriftenartikel der lizenzierten Zeitschriften werden selbstverständlich in PubPharm nachgewiesen. Weiterführende Informationen zu den Lizenzen sowie eine komplette Liste aller Zeitschriften können im PubPharm-Blog (https://blogs.tu-braunschweig.de/pubpharm/lizenzen/) eingesehen werden.
Neben der Versorgung der Fachcommunity mit aktuellen Publikationen wurden auch durch Retro-Digitalisierung zahlreiche pharmaziehistorische Werke zugänglich gemacht: Es stehen über den Publikationsserver der Technischen Universität Braunschweig mehr als 1.100 Bücher und 700 Zeitschriftenbände, überwiegend aus dem 19. und 20. Jahrhundert, frei zur Verfügung (z.B. „Pharmazeutische Zeitung“, „Deutsche Apotheker Zeitung”, „Pharmaceutical Historian“) [4].
Die Langzeitarchivierung, und damit die dauerhafte Sicherung und Zugänglichkeit der elektronischen Ressourcen, erfolgt in Kooperation mit einem nationalen Langzeitarchivierungszentrum an der TIB – Leibniz-Informationszentrum Technik und Naturwissenschaften. Der FID Pharmazie ist darüber hinaus im Bereich des fachspezifischen Forschungsdatenmanagements für die Community beratend tätig und wird – Bewilligung des Antrags vorausgesetzt – innerhalb der Nationalen Forschungsdateninfrastruktur für die Chemie (NFDI4Chem) pharmaziespezifische Bedarfe abdecken [5]. Die Services des FID Pharmazie werden in kontinuierlichem Austausch mit der pharmazeutischen Fachcommunity entwickelt und optimiert, so dass deren Anforderungen maßgeschneidert adressiert werden können. Dabei werden alle Teilbereiche dieses interdisziplinären Faches – Pharmazeutische Technologie, Pharmazeutische Biologie, Medizinische und Pharmazeutische Chemie, Pharmakologie und klinische Pharmazie sowie Pharmaziegeschichte – berücksichtigt. Ein Fachbeirat bestehend aus Wissenschaftler*innen der pharmazeutischen Teildisziplinen und der Informatik begleitet die Weiterentwicklung des FID Pharmazie.
Die Inhalte und Funktionalitäten von PubPharm und die weiteren Services des FID Pharmazie werden auf wissenschaftlichen Kongressen, Konferenzen und Jahrestagungen pharmazeutischer und bibliothekarischer Fachgesellschaften vorgestellt sowie dem wissenschaftlichen Nachwuchs an den verschiedenen Pharmaziestandorten in Roadshows präsentiert. Aktuelle Informationen aus dem FID Pharmazie und zur Weiterentwicklung der PubPharm-Funktionalitäten werden getwittert und im PubPharm-Blog sowie LinkedIn-Account gepostet.
PubPharm – Rechercheplattform für die Pharmazie
PubPharm ist ein auf der Open Source Software VuFind basierendes Discovery System [6]. Die Architektur besteht aus Datenquellen, Datenmanagement und Frontend. Als Recherchegrundlage enthält PubPharm bibliographische Daten und für die Rechercheunterstützung Normdaten, u.a. aus der Wirkstoffdatenbank DrugBank und den Medical Subject Headings (MeSH), um z.B. Suchvorschläge zu generieren [7], [8]. Die bibliographischen Daten aus verschiedenen Quellen werden im K10plus-Zentral der Verbundzentrale des GBV zusammengeführt, um sie integriert und performant durchsuchbar zu machen [9].
PubPharm enthält aktuell über 55 Millionen Nachweise. Dazu gehören 29 Millionen Publikationen aus der biomedizinischen Datenbank Medline/PubMed, weitere Artikel aus Fachzeitschriften angrenzender Disziplinen der Pharmazie (u.a. der Chemie, Pharmakologie oder auch Toxikologie), fachspezifische Bücher (z.B. E-Books und Dissertationen), Konferenzschriften und Informationen zu klinischen Studien aus dem Studienregister ClinicalTrials.gov [10], [11]. Inhaltlich liegt der Mehrwert von PubPharm damit in einem umfassenden Nachweis von unterschiedlichen Ressourcen, der quantitativ weit über die im Fach etablierten Datenbanken hinausgeht. PubPharm ist daher grundsätzlich nicht nur für die pharmazeutische Forschung, sondern auch für andere Lebenswissenschaften interessant und nützlich.
PubPharm ist frei verfügbar. Durch das responsive Design der Rechercheoberfläche ist das Arbeiten mit mobilen Endgeräten bequem möglich. Die Suche mit Booleschen Operatoren (AND, OR, NOT) wird ebenso unterstützt wie die Trunkierung (* als Platzhalter für eine beliebige Anzahl an Zeichen bzw. ? für ein einziges Zeichen). Für komplexere und selektivere Suchanfragen, z.B. bei der direkten Suche nach einem Zeitschriftentitel, kann die erweiterte Suche verwendet werden. Filterfunktionen (nach Medienart, Zeitschriftentitel, Thema, Erscheinungsjahr und Sprache sowie für die Eingrenzung auf klinische Studien aus dem Studienregister ClinicalTrials.gov) und die Änderung der Standardsortierung von „Neueste Zuerst“ nach „Relevanz“ oder „Älteste Zuerst“ ermöglichen Treffer einer Suchanfrage einzugrenzen sowie die Suchanfrage zu verfeinern. Des Weiteren können Suchergebnisse nach Verfasser*in und Zeitschriftentitel sortiert werden. Durch das Anfügen einer Tilde (~) und eines numerischen Wertes an eine Suchphrase (”...“) ist eine Wortabstandssuche möglich (z.B. „pharmaceutical technology“~2). Einzelne Treffer können als sogenannte Favoriten markiert und über die Favoritenliste in verschiedenen Formaten (z.B. EndNote, BibTex, MARC) heruntergeladen, als Linkliste per E Mail versendet, im Benutzerkonto gespeichert oder in ein Literaturverwaltungsprogramm exportiert werden. Die textbasierte Suche wird in PubPharm durch die Suche nach chemischen Strukturen ergänzt. Diese basiert auf der Datenbank PubChem [12]. Chemische Verbindungen werden in einen integrierten Editor eingezeichnet oder importiert (Abbildung 1 [Abb. 1]).
Abbildung 1: Die PubPharm-Struktursuche bietet die Identifikation von chemischen Verbindungen anhand der Struktur, SMILES/InChI-Code und des Substanznamens mit der Möglichkeit einer Ähnlichkeits- und Substruktursuche.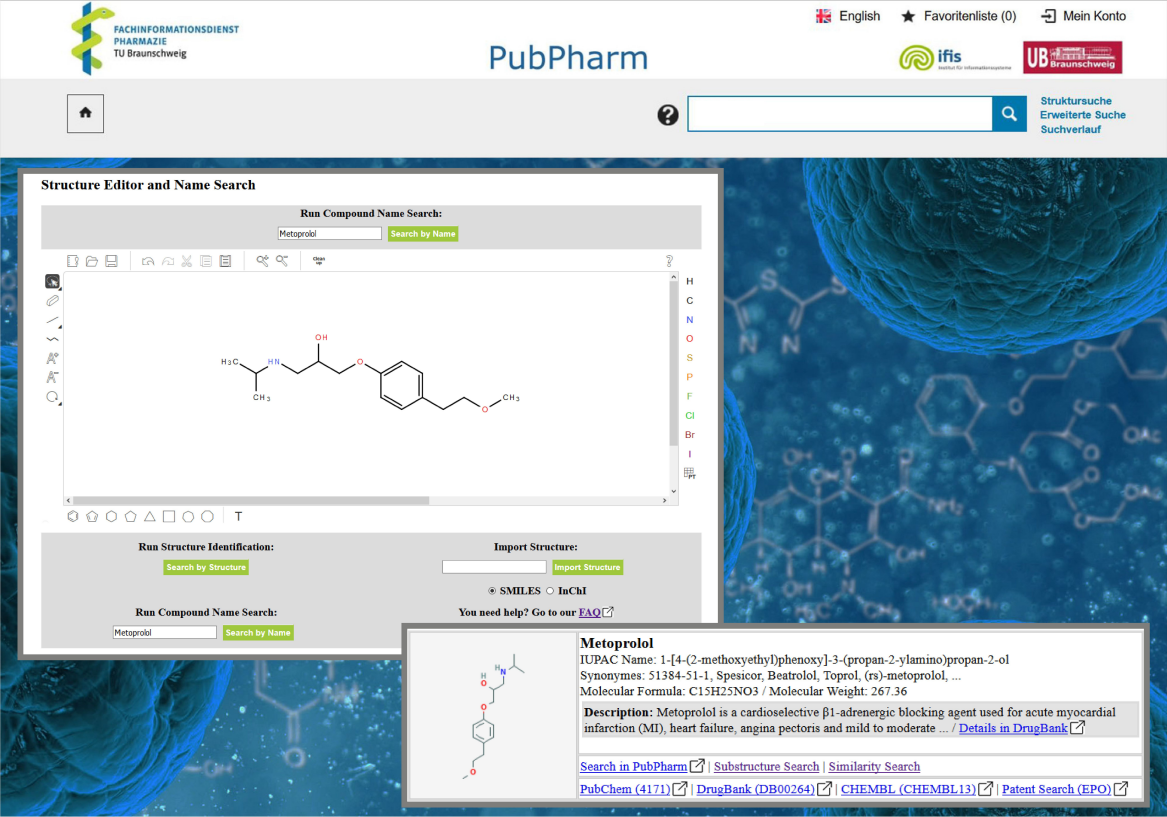
Zudem können Verbindungen anhand einer Namenssuche gefunden werden, wobei deren Strukturen dann in den Editor geladen und dort modifiziert werden können. Die Trefferübersicht enthält Informationen über die identifizierte Verbindung aus PubChem und, sofern verfügbar, der DrugBank. Weiterführende Informationen können über eine Literatursuche in PubPharm und Verlinkungen zur Suchoberfläche des Europäischen Patentamtes sowie zu den Datenbanken PubChem und ChEMBL erhalten werden [13], [14]. Die Struktursuche ist daher auch für die Suche nach Fakten zu einem Wirkstoff, u.a. zum Indikationsgebiet, Wirkmechanismus und nach Stoffeigenschaften, hilfreich. Zudem kann basierend auf PubChem Fingerprints zu jeder Verbindung nach Molekülen, die die Ausgangsstruktur enthalten und z.B. noch weitere Substituenten besitzen (Substruktursuche), oder mit nicht exakt übereinstimmenden Strukturelementen (Ähnlichkeitssuche), gesucht werden [15].
Die standortabhängige Verfügbarkeitsprüfung in PubPharm bietet den Nutzer*innen die Anzeige von Volltextzugriffsmöglichkeiten. Die Verfügbarkeitsprüfung erfolgt schrittweise. Auf Basis von bibliographischen Daten, die ggf. Digital Object Identifier (DOI) enthalten, und der Dienste „Journals Online & Print“ sowie „Unpaywall“ kann ermittelt werden, ob für eine Veröffentlichung eine frei zugängliche Version vorliegt [16]. Zusätzlich zur Abfrage nach der Verfügbarkeit einer elektronischen Lizenz prüft PubPharm auch die Verfügbarkeit von Druckbeständen in der jeweiligen Einrichtung des Nutzenden. Voraussetzung ist der korrekte Nachweis der Lizenzen in der EZB bzw. ZDB [17], [18]. Nachfolgend wird überprüft, ob eine FID-Lizenz vorliegt. Kann kein freier oder lizenzierter Zugriff auf einen Volltext ermittelt werden, bietet PubPharm die Verlinkung zum kostenpflichtigen Dokumentenlieferdienst subito an [19].
Ein Alleinstellungsmerkmal von PubPharm sind die innovativen Suchfunktionen, die vom Projektkooperationspartner, dem IfIS, passgenau erforscht und entwickelt wurden und werden. Der exponentielle Anstieg an Veröffentlichungen erfordert auch für das Fach Pharmazie neue Zugriffspfade zu Informationen, welche begriffsbasierte Suchen ergänzen können. Ein vom IfIS verfolgter Ansatz ist die automatische Extraktion von in Literatur eingebetteten pharmazeutischen Entitäten wie Wirkstoffen, Erkrankungen, Genen und deren Relationen untereinander. Künstliche neuronale Netze können in diesem Zusammenhang mit deutlichen Verbesserungen bezüglich Performanz, Skalierbarkeit und Genauigkeit existierende Relationen erlernen und sogar neue Relationen und Eigenschaften vorhersagen [20]. Die bisherigen Forschungsergebnisse konnten in einen Webservice überführt werden, der bereits in PubPharm implementiert ist. Dieser bietet Nutzer*innen Vorschlagslisten verwandter, kontextähnlicher Substanzen, Erkrankungen/Symptome und Gene bei der Suche nach einem Wirkstoff (Abbildung 2 [Abb. 2]), die z.B. bei der literaturbasierten wissenschaftlichen Hypothesenfindung und bei Untersuchungen zum Drug-Repositioning unterstützen können [21]. Die verwandten pharmazeutischen Entitäten werden alleine durch ihre semantische Ähnlichkeit zueinander identifiziert. Sie basieren nicht auf Strukturähnlichkeiten oder Therapieschemata. Netzwerkansichten auf Basis des Outputs künstlicher neuronaler Netze können die Interpretierbarkeit von komplexen Zusammenhängen vereinfachen [22], [23]. Am IfIS wurde bereits eine prototypische Anwendung entwickelt, auf deren Basis ein innovativer Service für PubPharm folgen wird.
Abbildung 2: Innovative Vorschlagsfunktion für die Suche nach dem Antibiotikum „Phenoxymethylpenicillin“. Es werden Vorschlagslisten mit kontextähnlichen, verwandten Substanzen generiert. Für den gesuchten Wirkstoff werden beispielsweise weitere Antibiotika (Cefaclor, Amoxicillin, Cefuroxim) und Erkrankungen, die in Abhängigkeit von der Ausprägung mit Antibiotika behandelt werden können, gelistet. 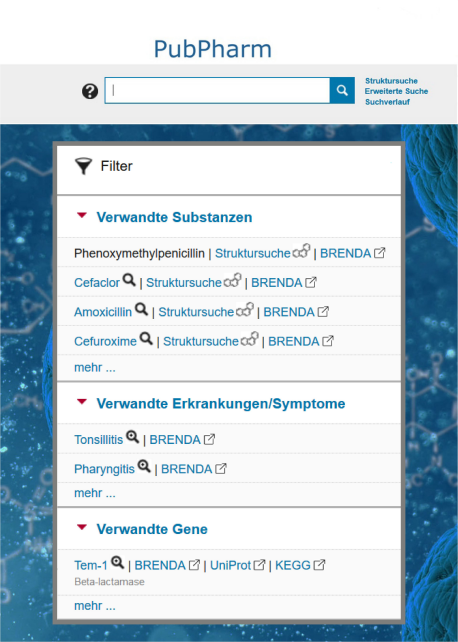
Fazit und Ausblick
Der FID Pharmazie hat mit substantieller Unterstützung durch die DFG forschungsunterstützende Services für wirkstoffbezogene Informationen und den unmittelbaren Zugriff auf pharmazierelevante Literatur aufgebaut. PubPharm konnte sich in der Fachcommunity als leistungsfähige Rechercheplattform etablieren, da es der Pharmazie einen maßgeschneiderten Zugang zu Informationsressourcen bietet, die vergleichbare Suchportale nicht bereitstellen. Alleinstellungsmerkmale wurden in stetiger Rückkopplung mit der pharmazeutischen Fachcommunity entwickelt, z.B. die Anzeige verwandter Substanzen, Erkrankungen/Symptome und Gene bei der Wirkstoffsuche. Aktuell werden neue Services erforscht und entwickelt: So untersucht das IfIS, ob sich die bereits etablierte Facettierungs-Methode erweitern lässt, um Prognosen für potentiell neue Indikationen oder Nebenwirkungen zu treffen. Zugleich werden die Methoden zur kontextbasierten Suche künftig auf Patente ausgeweitet. Dazu werden die Metadaten pharmazierelevanter Patente in PubPharm integriert. Zusätzliche Datenquellen, die in Kürze in den K10plus-Zentral Index übernommen und damit in PubPharm auffindbar werden, sind Preprint-Repositorien, z.B. bioRxiv und ChemRxiv, ein fachlicher Ausschnitt aus BASE sowie weitere Register klinischer Studien. Mittelfristig wird der Suchraum von PubPharm auch auf Forschungsdaten ausgeweitet. Zugleich werden zusätzliche Services im Bereich Forschungsdatenmanagement durch den FID Pharmazie etabliert. Entsprechende Bedarfe wurden in engem Kontakt mit der Fachcommunity bereits ermittelt.
Selbstverständlich ist der FID Pharmazie auch am Feedback angrenzender Fachbereiche, insbesondere in den Lebenswissenschaften, interessiert. Der FID Pharmazie ist daher auch auf einschlägigen Kongressen, Konferenzen sowie Tagungen präsent, um seinen zentralen Service PubPharm vorzustellen und Rückmeldungen einzuholen.
Anmerkung
Interessenkonflikte
Die Autoren erklären, dass sie keine Interessenkonflikte in Zusammenhang mit diesem Artikel haben.
Literatur
[1] Stump K, Balke WT, Keßler K, Krüger AT, Wawrzinek J, Wulle S. PubPharm – Der Fachinformationsdienst Pharmazie. Pharmakon. 2018;6(4):260-7.[2] Balke WT, Keßler K, Krüger AT, Stump K, Wawrzinek J, Wulle S. FID Pharmazie: Zwischen Spitzenforschung und verlässlicher Infrastruktur. ZfBB – Zeitschrift für Bibliothekswesen und Bibliographie. 2018;65(2-3):114-7.
[3] Wulle S. Das DFG-Sondersammelgebiet Pharmazie der Universitätsbibliothek Braunschweig. GMS Medizin – Bibliothek – Information. 2011;11(1-2):Doc08. DOI: 10.3205/mbi000223
[4] Universitätsbibliothek Braunschweig. Digitale Bibliothek: Der Publikationsserver der TU Braunschweig – Sammlung Pharmazie [Internet]. [updated 2019; cited 2019 Mar 25]. Available from: https://www.publikationsserver.tu-braunschweig.de/content/collections/pharmacy.xml
[5] NFDI4Chem – Chemistry Consortium in the NFDI [Internet]. [updated 2019; cited 2019 Nov 7]. Available from: https://www.nfdi4chem.de/
[6] vufind [Internet]. [updated 2019 Oct 11; cited 2019 Oct 24]. Available from: https://vufind.org
[7] DrugBank [Internet]. [updated 2019; cited 2019 Oct 25]. Available from: https://www.drugbank.ca/
[8] U.S. National Library of Medicine. Medical Subject Headings [Internet]. [updated 2019 Feb 23; cited 2019 Mar 25]. Available from: https://www.nlm.nih.gov/mesh/meshhome.html
[9] Verbundzentrale des GBV. K10plus-Zentral [Internet]. [updated 2019; cited 2019 Nov 7]. Available from: https://verbundwiki.gbv.de/display/VZG/K10plus-Zentral
[10] PubMed [Internet]. [updated 2019; cited 2019 Oct 24]. Available from: https://www.ncbi.nlm.nih.gov/pubmed/
[11] ClinicalTrials.gov [Internet]. [updated 2019; cited 2019 Oct 25]. Available from: https://www.clinicaltrials.gov/
[12] National Center for Biotechnology Information. The PubChem Project [Internet]. [updated 2019; cited 2019 Mar 25]. Available from: https://pubchem.ncbi.nlm.nih.gov/
[13] European Patent Office [Internet]. [updated 2019; cited 2019 Oct 25]. Available from: https://www.epo.org/index_de.html.
[14] ChEMBL [Internet]. [updated 2019; cited 2019 Oct 25]. Available from: https://www.ebi.ac.uk/chembl/
[15] National Center for Biotechnology Information. PubChem Substructure Fingerprint [Internet]. [updated 2018 May 18; cited 2019 Oct 25]. Available from: ftp://ftp.ncbi.nlm.nih.gov/pubchem/specifications/pubchem_fingerprints.pdf
[16] Journals Online & Print [Internet]. [updated 2019 Mar 20; cited 2019 Nov 7]. Available from: https://www.zeitschriftendatenbank.de/services/schnittstellen/journals-online-print/
[17] Elektronische Zeitschriftenbibliothek [Internet]. [updated 2019; cited 2019 Oct 24]. Available from: https://rzblx1.uni-regensburg.de/ezeit/
[18] Elektronische Zeitschriftendatenbank [Internet]. [updated 2019 Jan 28; cited 2019 Oct 24]. Available from: https://www.zeitschriftendatenbank.de/startseite/
[19] subito. Dokumente aus Bibliotheken e.V. [Internet]. [updated 2019; cited 2019 Oct 24]. Available from: https://www.subito-doc.de/
[20] Wawrzinek J, Balke WT. Semantic Facettation in Pharmaceutical Collections Using Deep Learning for Active Substance Contextualization. In: Choemprayong S, Crestani F, Cunningham S, editors. Digital Libraries: Data, Information, and Knowledge for Digital Lives. 19th International Conference on Asia-Pacific Digital Libraries (ICADL 2017); 2017 Nov 13-15; Bangkok, Thailand. Cham: Springer; 2017. (Lecture Notes in Computer Science; 10647). p. 41-53. DOI: 10.1007/978-3-319-70232-2_4
[21] Ashburn TT, Thor KB. Drug repositioning: Identifying and developing new uses for existing drugs. Nature reviews. Drug discovery. 2004;3(8):673-83.
[22] Wawrzinek J, Balke WT. Measuring the Semantic World – How to Map Meaning to High-Dimensional Entity Clusters in PubMed? In: Dobreva M, Hinze A, Žumer M, editors. Maturity and Innovation in Digital Libraries. 20th International Conference on Asia-Pacific Digital Libraries (ICADL 2018); 2018 Nov 19-22; Hamilton, New Zealand. Cham: Springer; 2018. (Lecture Notes in Computer Science; 11279). p.15-27. DOI: 10.1007/978-3-030-04257-8_2
[23] Wawrzinek J, Pinto JMG, Balke WT. Linking Semantic Fingerprints of Literature – from Simple Neural Embeddings Towards Contextualized Pharmaceutical Networks. In: 23rd International Conference on Theory and Practice of Digital Libraries (TPDL 2019); 2019 Sep 9-12; Oslo, Norway.




